-
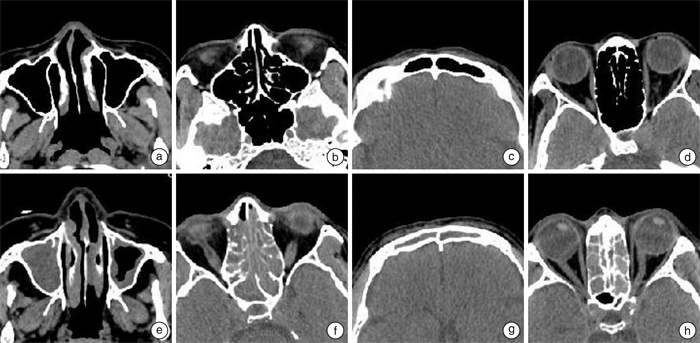
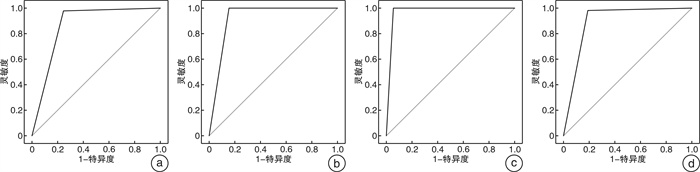
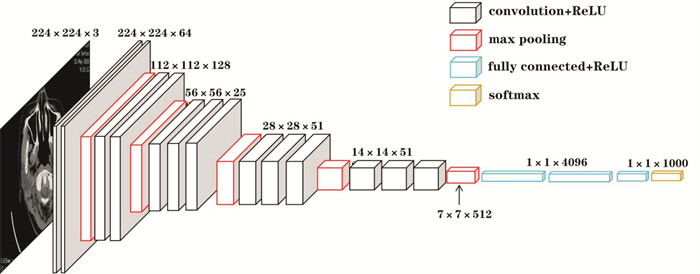
摘要: 目的 搭建基于VGG的慢性鼻窦炎计算机辅助诊断模型,并评价其效能。 方法 ① 收集5 000帧已确诊的鼻窦CT图像,将其分为正常组1 000帧图像(其中,正常的上颌窦、额窦、筛窦、蝶窦影像图像各250帧)及异常组4 000帧图像(其中,上颌窦炎、额窦炎、筛窦炎、蝶窦炎影像图像各1 000帧),对图像进行大小归一化及分割预处理;②训练模型并对其进行仿真实验,分别得到正常组,蝶窦炎组,额窦炎组,筛窦炎组以及上颌窦炎组5个分类模型,从准确度、精确度、灵敏度、特异度、判读时间及ROC曲线下面积(AUC)6个维度,客观评价模型的分类效能;③随机选取200帧图像,通过模型与低年资医师组、中年资医师组、高年资医师组分别阅片构成对比试验,结合临床通过以上评价指标客观评价模型的效能。 结果 ① 仿真实验:整个模型的识别准确度为83.94%,精确度为89.52%,灵敏度为83.94%,特异度为95.99%,平均每帧图像判读时间为0.20 s;蝶窦炎的AUC为0.865(95%CI 0.849~0.881),额窦炎的AUC为0.924(0.911~0.936),筛窦炎的AUC为0.895(0.880~0.909),上颌窦炎的AUC为0.974(0.967~0.982)。②对比实验:在识别准确度上,模型为84.52%,低年资医师组为78.5%、中年资医师组为80.5%,高年资医师组为83.5%;在识别精确度上,模型为85.67%,低年资医师组为79.72%,中年资医师组为82.67%,高年资医师组为83.66%;在识别灵敏度上,模型为84.52%,低年资医师组为78.50%,中年资医师组为80.50%,高年资医师组为83.50%;在识别特异度上,模型为96.58%,低年资医师组为94.63%,中年资医师组为95.13%,高年资医师组为95.88%;在耗时上,模型平均每帧图像为0.20 s,低年资医师组平均每帧图像为2.35 s,中年资医师组平均每帧图像为1.98 s,高年资医师组平均每帧图像为2.19 s。 结论 本研究强调了基于深度学习的慢性鼻窦炎人工智能诊断模型分类诊断慢性鼻窦炎的可能性;基于深度学习的慢性鼻窦炎人工智能诊断模型分类性能好,具有较高的诊断效能。Abstract: Objective To build a VGG-based computer-aided diagnostic model for chronic sinusitis and evaluate its efficacy. Methods ① A total of 5 000 frames of diagnosed sinus CT images were collected. The normal group consisted of 1 000 frames(250 frames each of maxillary sinus, frontal sinus, septal sinus, and pterygoid sinus), while the abnormal group consisted of 4 000 frames(1 000 frames each of maxillary sinusitis, frontal sinusitis, septal sinusitis, and pterygoid sinusitis). ②The models were trained and simulated to obtain five classification models for the normal group, the pteroid sinusitis group, the frontal sinusitis group, the septal sinusitis group and the maxillary sinusitis group, respectively. The classification efficacy of the models was evaluated objectively in six dimensions: accuracy, precision, sensitivity, specificity, interpretation time and area under the ROC curve(AUC). ③Two hundred randomly selected images were read by the model with three groups of physicians(low, middle and high seniority) to constitute a comparative experiment. The efficacy of the model was objectively evaluated using the aforementioned evaluation indexes in conjunction with clinical analysis. Results ① Simulation experiment: The overall recognition accuracy of the model is 83.94%, with a precision of 89.52%, sensitivity of 83.94%, specificity of 95.99%, and the average interpretation time of each frame is 0.2 s. The AUC for sphenoid sinusitis was 0.865(95%CI 0.849-0.881), for frontal sinusitis was 0.924(0.991-0.936), for ethmoidoid sinusitis was 0.895(0.880-0.909), and for maxillary sinusitis was 0.974(0.967-0.982). ②Comparison experiment: In terms of recognition accuracy, the model was 84.52%, while the low-seniority physicians group was 78.50%, the middle-seniority physicians group was 80.50%, and the seniority physicians group was 83.50%; In terms of recognition accuracy, the model was 85.67%, the low seniority physicians group was 79.72%, the middle seniority physicians group was 82.67%, and the high seniority physicians group was 83.66%. In terms of recognition sensitivity, the model was 84.52%, the low seniority group was 78.50%, the middle seniority group was 80.50%, and the high seniority group was 83.50%. In terms of recognition specificity, the model was 96.58%, the low-seniority physicians group was 94.63%, the middle-seniority physicians group was 95.13%, and the seniority physicians group was 95.88%. In terms of time consumption, the average image per frame of the model is 0.20 s, the average image per frame of the low-seniority physicians group is 2.35 s, the average image per frame of the middle-seniority physicians group is 1.98 s, and the average image per frame of the senior physicians group is 2.19 s. Conclusion This study demonstrates the potential of a deep learning-based artificial intelligence diagnostic model for chronic sinusitis to classify and diagnose chronic sinusitis; the deep learning-based artificial intelligence diagnosis model for chronic sinusitis has good classification performance and high diagnostic efficacy.
-
Key words:
- VGG /
- medical image /
- chronic rhinosinusitis /
- computer aided diagnosis
-
-
表 1 基于VGG的4组CRS分类模型混淆矩阵
帧 类型 蝶窦炎 额窦炎 筛窦炎 上颌窦炎 正常 蝶窦炎 678 30 169 42 81 额窦炎 0 820 2 2 176 筛窦炎 38 53 769 14 126 上颌窦炎 10 7 0 946 37 正常 1 0 10 5 984 表 2 准确率、精确率、灵敏度、特异度、判读时间评价结果
类型 ACC/% Precision/% SEN/% SPE/% 时间/s 蝶窦炎 93.26 67.80 98.78 额窦炎 90.11 82.00 97.75 筛窦炎 83.94 80.95 76.90 95.48 0.20 上颌窦炎 93.76 94.60 98.43 正常 70.09 98.40 89.50 表 3 不同年资医师组阅片混淆矩阵
帧 组别 蝶窦炎 额窦炎 筛窦炎 上颌窦炎 正常 低年资组 蝶窦炎 22 0 11 5 2 额窦炎 2 32 3 2 1 筛窦炎 4 1 33 1 1 上颌窦炎 1 1 2 35 1 正常 1 0 4 0 35 中年资组 蝶窦炎 23 0 10 7 0 额窦炎 2 34 4 0 0 筛窦炎 2 4 34 0 0 上颌窦炎 0 0 3 37 0 正常 1 1 4 1 33 高年资组 蝶窦炎 28 0 7 3 2 额窦炎 2 37 1 0 0 筛窦炎 2 4 31 1 2 上颌窦炎 0 0 0 35 5 正常 1 1 2 0 36 模型组 蝶窦炎 27 1 7 2 3 额窦炎 0 33 0 0 7 筛窦炎 2 2 31 0 5 上颌窦炎 0 0 0 39 1 正常 0 0 1 0 39 表 4 低年资医师组阅片评价
类型 ACC/% Precision/% SEN/% SPE/% 时间/s 蝶窦炎 73.33 55.00 95.00 额窦炎 94.12 80.00 98.75 筛窦炎 78.50 62.26 82.50 87.50 2.35 上颌窦炎 81.40 87.50 95.00 正常 87.50 87.50 96.88 AVE 78.50 79.72 78.50 94.63 2.35 表 5 中年资医师组阅片评价
类型 ACC/% Precision/% SEN/% SPE/% 时间/s 蝶窦炎 82.14 57.50 96.88 额窦炎 87.18 85.00 96.88 筛窦炎 80.50 61.82 85.00 86.88 1.98 上颌窦炎 82.22 92.50 95.00 正常 100.00 82.50 100.00 AVE 80.50 82.67 80.50 95.13 1.98 表 6 高年资医师组阅片评价
类型 ACC/% Precision/% SEN/% SPE/% 时间/s 蝶窦 84.85 70.00 96.88 额窦 88.10 92.50 96.88 筛窦 83.50 75.61 77.50 93.75 2.19 上颌窦 89.74 87.50 97.50 正常 80.00 90.00 94.38 AVE 83.50 83.66 83.50 95.88 2.19 表 7 模型阅片评价
类型 ACC/% Precision/% SEN/% SPE/% 时间/s 蝶窦 93.26 67.80 99.53 额窦 90.11 82.00 98.50 筛窦 84.52 79.69 76.90 96.20 0.2 上颌窦 95.21 97.50 98.80 正常 70.09 98.40 89.88 AVE 84.52 85.67 84.52 96.58 0.2 表 8 3个年资医师组及模型的分类AVE结果比较
组别 ACC/% precision/% SEN/% SPE/% 时间/s 模型组 84.52 85.67 84.52 96.58 0.20 低年资组 78.50 79.72 78.50 94.63 2.35 中年资组 80.50 82.67 80.50 95.13 1.98 高年资组 83.50 83.66 83.50 95.88 2.19 -
[1] 中华耳鼻咽喉头颈外科杂志编辑委员会鼻科组, 中华医学会耳鼻咽喉头颈外科学分会鼻科学组. 中国慢性鼻窦炎诊断和治疗指南(2018)[J]. 中华耳鼻咽喉头颈外科杂志, 2019, 54(2): 81-100.
[2] Liu Z, Chen J, Cheng L, et al. Chinese society of allergy and Chinese society of otorhinolaryngology-head and neck surgery guideline for chronic rhinosinusitis[J]. Allergy Asthma Immunol Res, 2020, 12(2): 176-237. doi: 10.4168/aair.2020.12.2.176
[3] 姚和梅, 刘领波, 李红英, 等. 慢性鼻窦炎及鼻息肉与性别和年龄的关系[J]. 中国耳鼻咽喉头颈外科, 2005(3): 183-184. https://www.cnki.com.cn/Article/CJFDTOTAL-EBYT200503023.htm
[4] 司马宇彤, 赵妍, 矫健, 等. 以慢性鼻窦炎内在型为导向的临床治疗选择[J]. 临床耳鼻咽喉头颈外科杂志, 2023, 37(11): 902-908. https://lceh.whuhzzs.com/article/doi/10.13201/j.issn.2096-7993.2023.11.009
[5] 赵愉, 王得旭, 顾力栩. 人工智能技术在计算机辅助诊断领域的发展新趋势[J]. 中国科学: 生命科学, 2020, 50(11): 1321-1334. https://www.cnki.com.cn/Article/CJFDTOTAL-JCXK202011014.htm
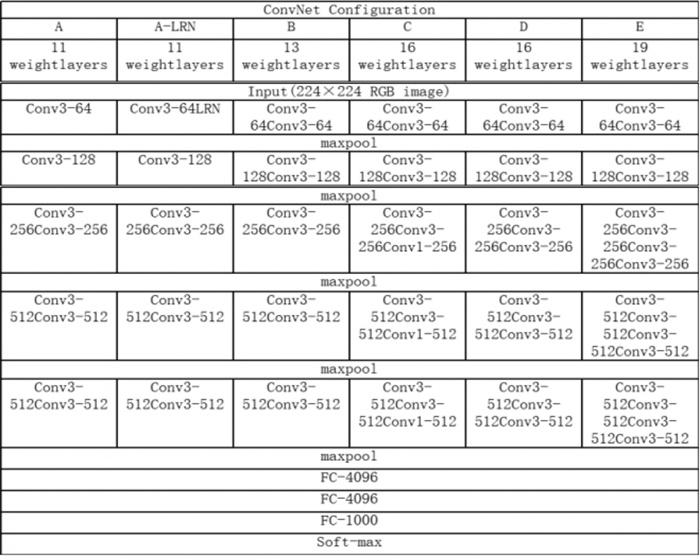
[6] Simonyan K, Zisserman A. Very deep convolutional networks for large-scale image recognition[J]. arXiv E Prints, 2014: arXiv: 1409.1556.
[7] Hong D, Zheng YY, Xin Y, et al. Genetic syndromes screening by facial recognition technology: VGG-16 screening model construction and evaluation[J]. Orphanet J Rare Dis, 2021, 16(1): 344. doi: 10.1186/s13023-021-01979-y
[8] Lin HX, Wei C, Wang GX, et al. Automated classification of hepatocellular carcinoma differentiation using multiphoton microscopy and deep learning[J]. J Biophotonics, 2019, 12(7): e201800435. doi: 10.1002/jbio.201800435
[9] Chowdhury NI, Smith TL, Chandra RK, et al. Automated classification of osteomeatal complex inflammation on computed tomography using convolutional neural networks[J]. Int Forum Allergy Rhinol, 2019, 9(1): 46-52. doi: 10.1002/alr.22196
[10] Kim Y, Lee KJ, Sunwoo L, et al. Deep learning in diagnosis of maxillary sinusitis using conventional radiography[J]. Invest Radiol, 2019, 54(1): 7-15. doi: 10.1097/RLI.0000000000000503
[11] Khan HA, Jue W, Mushtaq M, et al. Brain tumor classification in MRI image using convolutional neural network[J]. Math Biosci Eng, 2020, 17(5): 6203-6216. doi: 10.3934/mbe.2020328
[12] Romero-Lopez A, Giro-i-Nieto X, Burdick J, et al. Skin lesion classification from dermoscopic images using deep learning techniques[C]//Biomedical Engineering. Innsbruck, Austria. Calgary, AB, Canada: ACTAPRESS, 2017.
-
计量
- 文章访问数: 242
- 施引文献: 0



 下载:
下载: